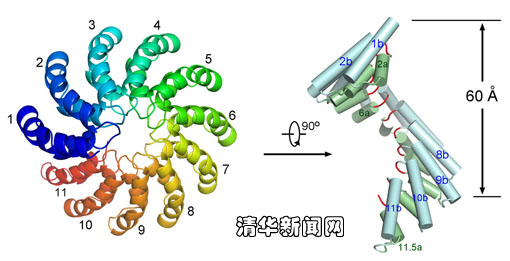
图为未结合DNA的dHax3晶体结构。
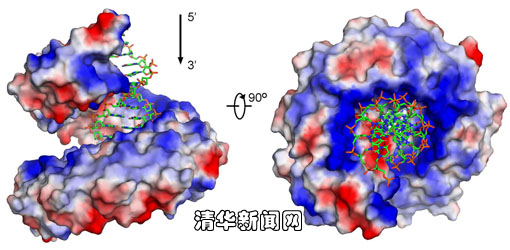
图为dHax3和DNA复合体的晶体结构。
2012年1月5日,清华大学颜宁教授研究组、施一公教授研究组以及美国普渡大学朱健康教授合作在《科学》在线发表论文,报道转录激活因子样效应蛋白(TALE)特异识别DNA的分子机理。
TALE (Transcription Activator Like Effectors)植物致病菌Xanthomonas通过III型分泌系统注入到宿主细胞内的一种蛋白质。TALE蛋白的奇特之处在于它的DNA结合结构域——该DNA结合结构域不同于其他已知的DNA结合结构域。它是由不同数量的重复单元组成,每一个重复单元特异识别一个DNA碱基对。大多数情况下每个重复单元由34个氨基酸组成。这34个氨基酸中除了第12,13位的氨基酸变化较大之外,其他氨基酸高度保守。这两个不保守的氨基酸被命名为RVD(repeat variable diresidue)。每个重复序列中12,13位的氨基酸和识别的核苷酸种类有特殊的一一对应关系。TALE蛋白的特异DNA序列识别以及灵活的可组装性为它们在分子生物学中的应用提供了巨大的前景,科学家们可以设计组装任意的TALE单元去识别目标DNA双螺旋序列。这一特性已经被用来构造切割特异双链DNA序列的DNA酶TALEN (TALE nuclease),成功用于在细胞基因组中引入定点突变、定点敲除等操作。理解TALE识别DNA的分子机制,会极大地促进其在 领域的应用。
TALE蛋白到底是如何实现这种特殊的DNA识别方式呢?为了回答这个有趣的问题,颜宁、施一公、朱健康研究组合作,选择了一个经过改造的TALE蛋白dHax3,进行结构生物学和生物化学研究,最终获得了2.4埃和1.85埃两个高分辨率的晶体结构:未结合DNA和结合DNA的TALE蛋白结构。这些晶体结构显示TALE蛋白的重复单元组成Helix-loop-helix的结构围绕DNA呈右手螺旋状排列,并清晰揭示了TALE蛋白特异识别DNA的机理。结构还显示RVD这两个残基中只有第二位的氨基酸才与碱基特异识别。结构比较进一步展示了TALE蛋白类似于弹簧的伸展性。这些结构信息提供了TALE蛋白的改造基础,极大地拓宽了TALE蛋白在生物技术应用上的前景。该论文以Highlight的形式被
Science Express发表。
该论文的共同第一作者是来自“清华、北大、NIBS三校联合培养项目”的二年级研究生邓东、清华大学生命学院的研究生闫创业,和清华大学医学院的一年级研究生潘孝敬。上海同步辐射(SSRF)为数据收集提供的及时有效的巨大便利保证了我国研究组在这一课题的激烈国际竞争中的最终胜出。(来源:清华大学 学院)
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。