|
|
|
|
|
快速诊断作物真菌病害的便携系统 | BMC Biology |
|
|
论文标题:MARPLE, a point-of-care, strain-level disease diagnostics and surveillance tool for complex fungal pathogens
期刊:BMC Biology
作者:Guru V. Radhakrishnan, Nicola M. Cook, […] Diane G. O. Saunders
发表时间:2019/08/13
DOI:10.1186/s12915-019-0684-y
微信链接:https://mp.weixin.qq.com/s/7a1xU39f4A-saQ5MSXqsBA
植物病虫害是全球农业和粮食安全面临的重大问题。早期预警系统对准备和抗击植物害虫至关重要,而快速诊断工具对这一点至关重要。然而,目前用于菌株水平的真菌疾病诊断程序依旧是冗长而不直接的。Dave Hodson和Diane Saunders讨论了他们近期发表在BMC Biology 上的一项研究,该研究开发了一种移动实时植物病害(Mobile And Real-time PLant disEase,MARPLE)系统。在接下来的分享中,他们解释了该系统如何使基于田间基因的真菌病害快速诊断成为可能,特别是判别达到菌株水平的小麦黄锈病。

埃塞俄比亚小麦叶上的锈斑
图片来源:Matt Heaton, John Innes Centre
植物病虫害是全球粮食安全的一大威胁,如果不加以处理,有可能毁掉整个作物。据估计,仅在前五大粮食作物中阻止真菌疾病的传播就可以养活6亿多人。小麦是全球五大农作物之一,目前这种作物正面临越来越大的威胁,其中真菌病原体引起的小麦锈病是最令人担忧的问题之一。
如今,由于无法获得有效的诊断试验和控制措施,欠发达国家的作物遭受大规模损失的风险最大。就像诊疗人类疾病一样,如果你看到明显的症状,但不确定病因,如何才能采取正确的治疗措施?而这,就是基于基因组的诊断和疾病监测可以发挥作用的地方。尤其是对于相关资源较少的国家,确定病原体的精确毒株,确保采取最适当的控制措施,有助于更明智地使用有限的资源。
技术革新:便携式基因组测序设备助力人类疾病诊断
在临床实验室中,研究人员经常解开人类疾病病原体的完整遗传密码来协助疾病诊断。自2015 Oxford Nanopore发布了首个便携式基因组测序仪以来,这种基因组水平的诊断和监测如今已经可以精确地在疾病暴发的地点进行,无论那里的环境多么不适宜人类居住。这一技术革新彻底改变了诊断的速度,建立了关于病原体菌株进化速度的实时基因组信息,并帮助绘制了它们的传播路线。

Dave Hodson博士与埃塞俄比亚农民合作,收集受锈病感染的黄色小麦样本
图片来源:Matt Heaton, John Innes Centre
这些最初的研究集中在病毒病原体上,但其实,对真菌疾病的实时基因组水平诊断也是至关重要且迫切被需要的。遗憾的是,对真菌使用这样的诊断策略并不简单。真菌基因组通常比病毒大数万倍,而且往往无法从宿主植物中分离出来。我们不能仅仅对含有真菌和(相当大的)寄主植物基因组的受感染叶片进行测序,因为基于目前的技术这将是非常昂贵的。我们需要一种不同的方法。
因“植”制宜:实现基于便携式基因组的真菌病原体诊断
我们决定使用一种叫做“靶向重测序”的方法,这种方法经常被用于测试特定的人类疾病。这种方法的重点是分析一小部分基因的遗传密码,这些基因可以提供特定条件的信息。这种有针对性的方法大大减少了每个样本生成的数据量,从而降低了相关的成本。
在近期发表于BMC Biology 的研究中,我们重点研究了小麦黄锈病真菌病原体(Puccinia striiformis f.sp. tritici)。我们首先确定了在病原体菌株之间的DNA代码中表现出高变异性的基因,然后使用这一小部分基因进行靶向重测序。这使我们能够快速确定受感染的叶片样本中的特定菌株。这个新的系统被称为MARPLE(Mobile And Real-time PLant disEase,移动实时植物病害)系统,它也非常适合于突发的病害事件,因为它可以在收集样本后的48小时内生成该农田内的黄锈病病原体菌株的详细数据。
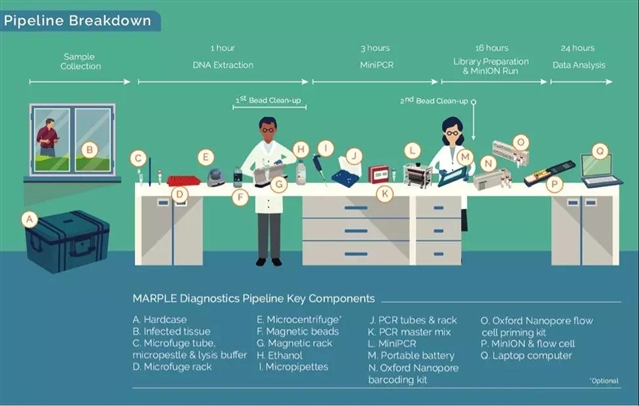
MARPLE系统(A-Q)的完整步骤示意图
MARPLE:为埃塞俄比亚小麦带去希望
埃塞俄比亚是撒哈拉以南非洲地区最大的小麦生产国,但它也是新小麦黄锈病病原体株进入非洲的门户,因此是实施快速诊断计划的最高优先国家。小麦黄锈病持续困扰着埃塞俄比亚的小麦生产,并造成了大规模的植物流行病。例如,2010年,60万公顷小麦受到影响,估计损失超过2.5亿美元,而新的病原体菌株对再次发生大规模流行病的风险非常高。
在如此大规模的破坏之后,埃塞俄比亚开发了一个综合小麦锈病早期预警系统,以防止未来灾难性的损失。然而,用目前常见的方法,在农民的田里实时确定病原体的精确品系是不可能的。MARPLE的设计正是为了填补这一空白。
与埃塞俄比亚农业研究所(Ethiopian Institute for Agricultural Research,EIAR)合作,我们开发了一个移动实验室系统,它被安置在一个独立的箱子中,不需要额外的基础设施或高水平的专业知识就可以操作。2018年9月,我们在埃塞俄比亚成功试运行了优化后的MARPLE系统。作为其稳健性和机动性的最终证明,我们还在一辆陆地巡洋舰的后备箱里直接在田野现场运行了这一系统,证明了无需连续供电或使用任何额外实验室设备操作的可行性。
Insert fig 4
Diane Saunders博士和EIAR的科学家们在埃塞俄比亚田边的一辆陆地巡洋舰后备箱操作执行MARPLE系统
图片来源:Matt Heaton, John Innes Centre
我们在John Innes Centre和CIMMYT之间的合作伙伴关系也允许直接将由这种方法产生的数据用于预警系统和控制建议体系中。该项目的整合目前使埃塞俄比亚成为小麦锈病病原体诊断和预测方面的世界领导者。
MARPLE的未来发展
我们为目标重测序开发的基因面板也非常灵活,可以根据需要轻松容纳额外的基因。目前,我们正在整合编码蛋白质的基因,这些蛋白质被认为是真菌病原体中保守的杀菌剂靶点。这将允许实时突变监测,从而提醒我们对这种黄锈病病原体的杀菌剂效用进行调整突破。MARPLE系统为可移动的、基于基因组的、菌株水平诊断提供了有迹可寻的图景,并可以很容易地应用于持续威胁全球粮食安全的其他复杂真菌病原体。
摘要:
Background
Effective disease management depends on timely and accurate diagnosis to guide control measures. The capacity to distinguish between individuals in a pathogen population with specific properties such as fungicide resistance, toxin production and virulence profiles is often essential to inform disease management approaches. The genomics revolution has led to technologies that can rapidly produce high-resolution genotypic information to define individual variants of a pathogen species. However, their application to complex fungal pathogens has remained limited due to the frequent inability to culture these pathogens in the absence of their host and their large genome sizes.
Results
Here, we describe the development of Mobile And Real-time PLant disEase (MARPLE) diagnostics, a portable, genomics-based, point-of-care approach specifically tailored to identify individual strains of complex fungal plant pathogens. We used targeted sequencing to overcome limitations associated with the size of fungal genomes and their often obligately biotrophic nature. Focusing on the wheat yellow rust pathogen, Puccinia striiformis f.sp. tritici (Pst), we demonstrate that our approach can be used to rapidly define individual strains, assign strains to distinct genetic lineages that have been shown to correlate tightly with their virulence profiles and monitor genes of importance.
Conclusions
MARPLE diagnostics enables rapid identification of individual pathogen strains and has the potential to monitor those with specific properties such as fungicide resistance directly from field-collected infected plant tissue in situ. Generating results within 48 h of field sampling, this new strategy has far-reaching implications for tracking plant health threat
(来源:科学网)
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。