|
|
|
|
|
FCS | 文章解读:FragDPI——基于片段理解和统一编码的新型药物-蛋白质相互作用预测模型 |
|
|
论文标题:FragDPI: a novel drug-protein interaction prediction model based on fragment understanding and unified coding(FragDPI——基于片段理解和统一编码的新型药物-蛋白质相互作用预测模型)
期刊:Frontiers of Computer Science
作者:Zhihui YANG, Juan LIU, Xuekai ZHU, Feng YANG, Qiang ZHANG, Hayat Ali SHAH
发表时间:15 Oct 2023
DOI:10.1007/s11704-022-2163-9
微信链接:点击此处阅读微信文章
原文信息
标 题:
FragDPI: a novel drug-protein interaction prediction model based on fragment understanding and unified coding
发表年份:
2023年
原文链接:
https://journal.hep.com.cn/fcs/EN/10.1007/s11704-022-2163-9
引用格式:
Zhihui YANG, Juan LIU, Xuekai ZHU, Feng YANG, Qiang ZHANG, Hayat Ali SHAH. FragDPI: a novel drug-protein interaction prediction model based on fragment understanding and unified coding. Front. Comput. Sci., 2023, 17(5): 175903
公众号推文链接:
文章精要 | FragDPI: 基于片段理解和统一编码的新型药物-蛋白质相互作用预测模型
01 导读
药物-蛋白结合预测是虚拟药物筛选的关键,目前已有许多基于蛋白质序列和药物表示序列的深度学习方法来预测药物-蛋白质结合。然而,现有的方法大多是分别从蛋白质和药物序列中提取特征。因此,他们不能了解表征药物-蛋白质相互作用的特征。此外,现有的蛋白质(药物)序列编码方法通常基于每个氨基酸(原子)对结合的贡献相同的假设,忽略了不同氨基酸(原子)对结合的不同影响。然而,药物-蛋白结合事件通常发生在蛋白质序列中的保守残基片段和药物分子的原子片段之间。因此,需要一种更全面的编码策略来从保守片段中提取信息。
本文提出了一个新的模型,命名为FragDPI,以预测药物-蛋白质结合亲和力。与其他方法不同的是,FragDPI基于保守片段对序列进行编码,将蛋白质和药物编码成一个统一的载体。此外,本文还采用了一种新的两步训练策略来训练FragDPI,两个步骤分别为预训练步骤和微调步骤,预训练步骤是使用无监督学习来学习不同片段之间的相互作用,微调步骤是为了使用监督学习预测绑定亲和力。最后,本文实验结果表明了FragDPI算法的优越性。
02 方法介绍
综述
FragDPI是一种预测药物-蛋白结合亲和力的深度神经网络模型,本文将变压器编码器模块应用于序列片段,有助于了解蛋白质序列中保守片段与药物分子的相互作用。模型结构如图1所示,通过FCS算法得到蛋白质序列和药物SMILES下降频率的片段词汇表,在获得药物蛋白对的保守片段后,用词汇对片段进行标记。然后对标记后的片段进行嵌入操作,包括标记嵌入和位置嵌入,以获取序列文本信息和位置信息。接下来,得到药物蛋白对每个片段的嵌入载体,并相互作用模块,将药物包埋载体和蛋白质包埋载体连接在一起,探索两者的相互作用片段。拼接完成后,将嵌入向量放入双向变换编码器中。
FragDPI采用了一种新的两步训练策略,即片段理解的预训练阶段和预测结合亲和力的微调阶段,如图1所示。这两个阶段之间模型的不同之处在于,微调阶段模型中被添加了完全连接的层来输出亲和度分数,算法1概述了训练过程。
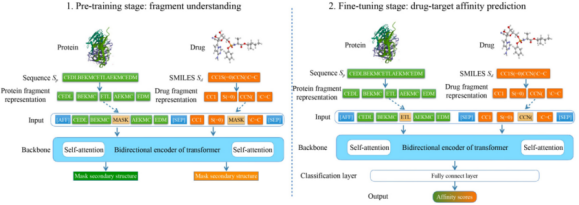
图1 模型概述(左侧为FU预训练阶段,右侧为微调阶段)

算法1
1.保守碎片开采
在对药物(蛋白质)序列进行标记之前,需要挖掘和识别保守片段以构建片段词汇表。由于缺乏标记子序列数据,本文使用FCS(频繁连续子序列挖掘算法)来识别蛋白质氨基酸序列和药物SMILES序列中的保守片段。
FCS算法根据大量未标记数据中标记的频率识别出一组相似的基本生化保守片段,扫描标记集并识别最频繁的连续标记,然后用新标记更新标记集中的每个标记。此操作将频繁子序列合并到一个标记中,而频率不够高的子序列被分解成更短的标记。FCS算法在大量蛋白质数据集中提供高频率的保守片段,帮助模型探索结合位置的功能基序。
使用FCS算法,本文得到了一个保守片段的词汇量为23,614。
2.统一的令牌编码
在保守片段挖掘后,根据保守片段的词汇表对蛋白质序列和药物SMILES进行标记。
为了了解药物蛋白对的保守片段之间的关系,本文用一个统一的编码向量作为模型的输入。因此,将药物和蛋白质序列串联起来,同时进行编码。如图2所示,将连接的序列拆分为保守片段,保守片段的标记化对应于在上一步中获得的词汇表索引。因此,得到了序列标记化,即包含每个保守片段的标记数的一维向量。保守片段词汇表按照长片段排在前面,短片段排在最后进行排序。
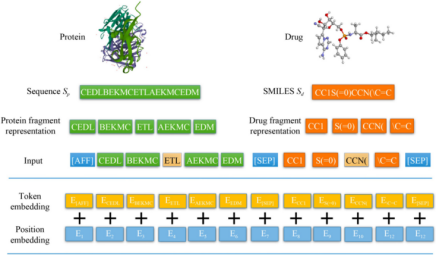
图2 模型的输入(包括标记嵌入和位置嵌入)
使用令牌嵌入表示保守片段的语义,使用位置嵌入提供保守片段的位置信息
1.嵌入模块
为了捕获保守片段的生物语义,将Vaswani A等人提出的编码方法应用于序列的标记向量。在序列建模任务中,现代研究利用令牌嵌入来表示序列的内容,利用位置嵌入来捕获序列中的位置信息。标记嵌入和位置嵌入是建立在具有随机初始化参数的查找字典上的,令牌嵌入和位置嵌入的蛋白质参数矩阵表示为 和
和 ,药物参数矩阵为
,药物参数矩阵为 和
和 ,最后的嵌入是标记嵌入和位置嵌入的总和。可以通过下面的方程得到药物表示
,最后的嵌入是标记嵌入和位置嵌入的总和。可以通过下面的方程得到药物表示 和蛋白质表示
和蛋白质表示 :
:

交互模块
本文选择双向变压器作为交互模块的骨干,大多数现代研究都是将药物和蛋白质分开编码,认为编码的过程应该在同一个领域。连接 和
和 馈送到交互模块,具体来说,模型的输入表示为E,表征为
馈送到交互模块,具体来说,模型的输入表示为E,表征为 (
( 表示连接操作),如图1中的“输入”所示。
表示连接操作),如图1中的“输入”所示。

FragDPI利用这种形式输入模型,可以模拟蛋白质与药物之间的保守片段之间的关系,双向变压器编码器利用自关注机制从其他片段的信息中建模隐藏状态。
片段的理解
为了使模型能够理解保守片段之间的对应关系,本文设计了一个无监督任务片段理解(Fragment Understanding, FU)。如图1所示,在药物和蛋白质输入中随机屏蔽片段,预训练的目标是预测被遮挡的片段。正式地说,整个输入序列有n个令牌 。此外,记录掩模的位置,
。此外,记录掩模的位置, 是最后一层隐藏状态的掩码位置。总之,预训练任务可以被格式化如下:
是最后一层隐藏状态的掩码位置。总之,预训练任务可以被格式化如下:

本文试图让模型理解片段的对应关系,将药物序列和蛋白质序列随机分别掩码。掩码策略为:在80%的概率下,随机掩码每个序列中15%的片段;将掩码令牌替换为特殊令牌,如图1所示。
DPI预测
微调任务是DPI预测,预测输入药物和蛋白质的亲和力评分。在对FU进行预训练后,使用预训练参数初始化模型。本文还增加了一个预测头来接受特殊令牌 的隐藏。具体来说,每个输入都将添加一个特殊的标记作为开始标记
的隐藏。具体来说,每个输入都将添加一个特殊的标记作为开始标记 ,该标记没有语义。此外,它还可以关注输入中的所有令牌,受自然语言处理中文本分类的启发,将
,该标记没有语义。此外,它还可以关注输入中的所有令牌,受自然语言处理中文本分类的启发,将 的隐藏状态记为
的隐藏状态记为 输入到预测头
输入到预测头 中,输出相应药物和蛋白质的得分。综上所述,该任务可以描述为以下形式:
中,输出相应药物和蛋白质的得分。综上所述,该任务可以描述为以下形式:

通常DPI预测被分为两个阶段的训练任务,第一阶段的任务是利用其他片段的信息来预测被屏蔽的片段,第二阶段,模型基于对碎片关系的理解进行DPI预测。
03 主要贡献
1.本文提出了一个深度学习模型,利用双向变形器作为主干,FragDPI来预测药物蛋白的结合亲和力。此外,还引入了一种统一的编码策略,通过识别序列中的保守片段来标记蛋白质序列。本文将药物和蛋白质编码成一个统一的载体, 以更精确地描述药物-蛋白质相互作用的细节,简化编码过程。
2.本文通过一种新的两步训练策略来构建模型。在预训练阶段,提出FU来探索蛋白质序列片段和药物分子片段之间的潜在信息;在微调阶段,使用预先训练好的FragDPI在特定的数据集中进行训练,然后完成药物-蛋白对的亲和力预测。
3.本文在四个数据集上使用最先进的方法进行广泛的性能评估。结果表明,FragDPI在RMSE和皮尔逊相关系数上达到了相当的性能。与同任务方法相比,FragDPI通过模型的注意力模块,可以从蛋白质序列与药物分子串之间的保守片段中挖掘出更多的结合特征。
04 实验结果总结
为了获得该任务的最优模型,本文在测试数据集上进行了参数网格实验,使用RMSE和皮尔逊相关系数作为指标来展示模型的性能。发现FragDPI在使用6层注意力块时表现最佳,每个注意力块有12个头,RMSE为0.84,皮尔逊相关系数为0.84,FragDPI的隐藏大小和嵌入大小为384。
05 与其他相关研究的对比
随着深度学习在包括生物信息学在内的许多领域的成功应用,最近提出了许多基于深度学习的DPI预测方法。一般来说,这些端到端方法直接从蛋白质和药物序列中提取特征,并据此预测药物-蛋白质的结合亲和力,因此,编码蛋白质(药物)序列对于构建模型至关重要。大多数现有方法通常使用独热向量来表示氨基酸(原子),然后使用独热矩阵来表示蛋白质(药物)序列。这种策略隐含地基于这样的假设,即单个残基(原子)是结合的功能单位,每个残基(原子)在药物-蛋白质结合中起着相同的作用。然而,在生化过程中,DPI通常发生在蛋白质和药物序列的保守片段之间,而不是单个残基和原子之间。显然,独热矩阵编码方法无法揭示实际药物-蛋白结合的细节。此外,大多数现有的方法采用两流网络模块,分别提取药物和蛋白质的特征。这种特征提取模块没有学习到药物-蛋白质相互作用的信息,缺乏对药物与蛋白质相互作用的局部研究。
而本文提出的名为FragDPI的新方法,用于预测药物-蛋白质结合亲和力。通过使用保守的片段而不是单个氨基酸或原子来编码蛋白质和药物,模拟了生化过程。采用统一的编码策略将药物和蛋白质的片段组合在一起,以提取相互作用的特征。并采用了一种新颖的两步训练策略来构建模型。
解读:戴西件 南昌大学第二附属医院
审核:张 琨 合肥工业大学

Frontiers of Computer Science
Frontiers of Computer Science (FCS)是由教育部主管、高等教育出版社和北京航空航天大学共同主办、SpringerNature 公司海外发行的英文学术期刊。本刊于 2007 年创刊,双月刊,全球发行。主要刊登计算机科学领域具有创新性的综述论文、研究论文等。本刊主编为周志华教授,共同主编为熊璋教授。编委会及青年 AE 团队由国内外知名学者及优秀青年学者组成。本刊被 SCI、Ei、DBLP、INSPEC、SCOPUS 和中国科学引文数据库(CSCD)核心库等收录,为 CCF 推荐期刊;两次入选“中国科技期刊国际影响力提升计划”;入选“第4届中国国际化精品科技期刊”;入选“中国科技期刊卓越行动计划项目”。
《前沿》系列英文学术期刊
由教育部主管、高等教育出版社主办的《前沿》(Frontiers)系列英文学术期刊,于2006年正式创刊,以网络版和印刷版向全球发行。系列期刊包括基础科学、
、工程技术和人文社会科学四个主题,是我国覆盖学科最广泛的英文学术期刊群,其中12种被SCI收录,其他也被A&HCI、Ei、MEDLINE或相应学科国际权威检索系统收录,具有一定的国际学术影响力。系列期刊采用在线优先出版方式,保证文章以最快速度发表。
中国学术前沿期刊网
http://journal.hep.com.cn

特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。