蛋白质功能注释是通过对蛋白质序列、结构的分析确定蛋白质功能,在生物学研究等领域具有重要意义。随着AlphaFold2、ESMFold等人工智能的方法开发,目前已经可以通过计算方法获得比较准确的蛋白结构,但从蛋白序列到功能的注释仍然面临着巨大挑战。目前,UniProt蛋白库(包含大约1.9亿个蛋白序列),只有不到0.3%(约50万个)经过了人工审核,其中仅有不到19.4%的蛋白质得到了明确的实验证据支持,这意味着蛋白质功能注释仍然高度依赖计算注释方法。酶号(EC)是国际酶学委员会制定的一套酶的编号分类法,从大类上将酶分成氧化还原酶、转移酶、水解酶、裂合酶、异构酶、连接酶等,通常由4个数字组成(比如EC 3.14.11.4),对于准确理解酶的功能和细胞代谢至关重要。目前已经提出了许多基于计算的方法来预测给定输入蛋白质序列的EC号,但这些方法在处理最近发现的蛋白质时,预测性能(准确度、召回率、精确度)、可用性和效率严重下降,仍有很大的改进空间。
为了解决这一问题,中国科学院天津工业生物技术研究所生物设计中心提出了一种名为Hierarchical Dual-core Multitask Learning Framework(简称“HDMLF”)的新型深度学习框架。HDMLF包含嵌入核和学习核,其中嵌入核利用最新的蛋白质语言模型对蛋白质序列进行嵌入表示,而学习核则用于EC号的预测。HDMLF以门控循环单元(GRU)为基础,以多目标层次、多任务的方式进行EC号预测。另外,还引入了注意力层对模型进行优化,并采用贪心策略集成和微调最终模型。与DeepEC等四种代表性方法进行的对比分析表明,HDMLF稳定地提供了最高的性能,准确度和F1分数分别提高了60%和40%。同时,相比较其他方法,HDMLF对于新数据集的预测有着显著优势。此外,该方法成功预测出大肠杆菌酪氨酸氨基转移酶tyrB的混杂性,准确预测tryB同样能催化天冬氨酸氨基转移酶aspC所催化的反应,展示了该方法在揭示酶的多样性方面的潜力。
为了提高该研究的可用性,研究团队同时发布了一个名为ECRECer的网站平台(https://ecrecer.biodesign.ac.cn),提供在线的蛋白注释服务,并提供离线工具,以提升用户的体验和可用性。总体而言,ECRECer是基于序列预测酶催化功能的强大工具,可以促进酶学、代谢工程、合成生物学等领域的研究。
该研究得到国家重点研发计划、国家自然基金委青年基金项目、中国博士后基金会面上项目、天津市合成生物技术创新能力提升行动、合成生物学海河实验室创新基金和中国科学院青年创新促进会等支持,相关成果发表在综合性期刊Research上。天津工业生物所博士后史振坤为论文的第一作者,廖小平副研究员和马红武研究员为论文的共同通讯作者。(来源:中国科学院天津工业生物技术研究所)
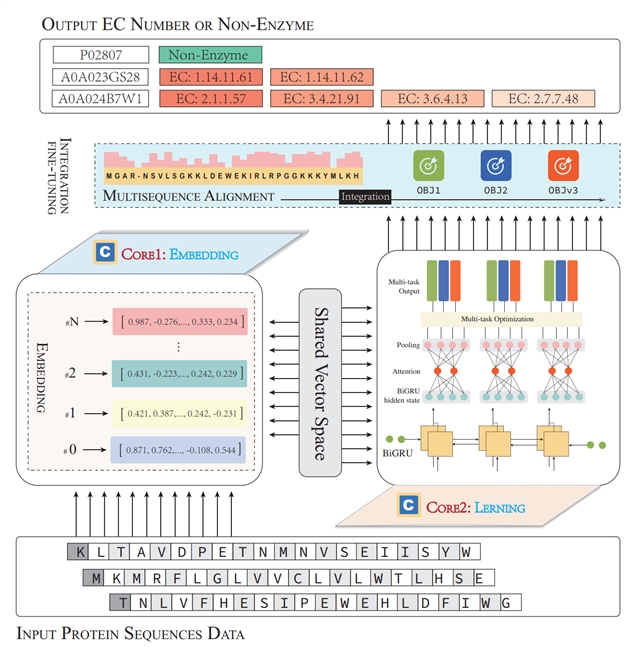
蛋白功能注释算法框架
相关论文信息:https://doi.org/10.34133/research.0153
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。