日前,国际知名机器学习学术期刊Nature Machine Intelligence(NMI,近2年影响因子≈16.649)在线发表了百度生物计算团队(“螺旋桨”团队)的最新研究成果,论文提出基于空间结构的化合物表征学习方法,即“几何构象增强AI算法”(GEM模型),将化合物的几何结构信息引入自监督学习和分子表示模型,对化合物进行“3D建模”,以预测化合物分子的性质属性。其在药物筛选中的应用,可在数小时内完成传统仿真方法1年的工作量,效率提升上千倍,有望大幅降低药物研发的时间投入和成本投入。
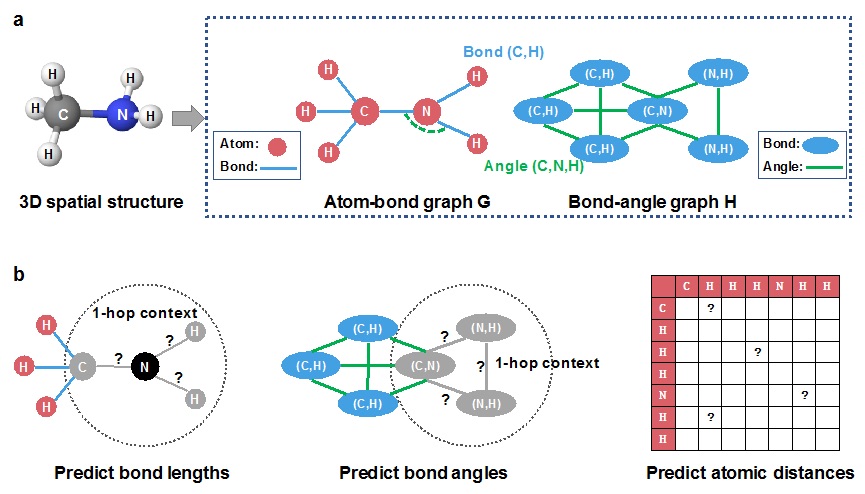
作为药物研发的关键一环,候选化合物的性质预测相当于为临床实验“排雷”,即提前筛选掉毒副作用高、人体不易吸收代谢等的化合物。此前,这项工作只能通过传统仿真实验进行,成本高且耗时长。后来,研究人员引入深度学习,但传统的深度学习方法大多基于序列或二维图结构建模,缺乏对化合物三维空间结构信息的利用,这会丢失一部分空间信息,导致化合物性质预测结果的偏差。为更好地预测化合物性质,亟需引入化合物的三维空间信息。
百度螺旋桨团队提出的GEM模型,正是在这一点上实现了突破。论文称,他们开创性的工作,是业界首次将化合物性质预测从“2D建模”推进到“3D建模”。同时,团队通过引入预训练技术和利用大量无标注的化合物数据,通过自监督学习来构建GEM模型的底层能力,有望打造小分子药物研发领域的模型底座,解决小分子药物活性预测、成药性预测等药物设计的核心问题,真正加速药物特别是创新药物的发现过程。
基于空间结构的化合物表征学习方法(GEM)的整体框架 百度供图
论文进一步发布了该模型的实验效果,结果显示,GEM模型在14个学术界公认的应用任务数据集(包括抑制 HIV 艾滋病病毒复制能力的数据集、小分子的生物活性数据集、血脑屏障渗透数据集等)上取得最佳结果,超越斯坦福大学等提出的模型效果。其中,GEM模型在“回归”任务上相对现有方法指标提升8.8%,在“分类”任务上指标相对提升4.7%,并在自监督学习方法上的消融实验中证明了其有效性。
目前,开源社区GitHub上已经开源了GEM模型完整代码。研究人员表示,GEM模型目前已经在多个合作伙伴的研发管线中实现了商业化落地。这表明,人们有望通过AI技术探索双靶点抑制剂新的研发范式,为癌症病人和自身免疫性疾病病人提供更有效的治疗药物。
此外,研究人员指出,该方法还有助于高效测量“药物—靶标”的相互作用,进而用于加速新药研发、发掘老药新用途、探索多种药物联合使用等,这有助于降低药品抗药性和毒副作用,甚至疗治新病症。
该项研究由百度螺旋桨PaddleHelix团队独立完成。百度基于飞桨打造的生物计算平台“螺旋桨PaddleHelix”,致力于为生物医药专家与学者提供“AI+生物计算”的模型工具和解决方案,服务于新药研发、疫苗设计、精准医疗等场景。(来源:中国科学报 赵广立)
相关论文信息:http://doi.org/10.1038/s42256-021-00438-4
版权声明:凡本网注明“来源:中国科学报、科学网、科学新闻杂志”的所有作品,网站转载,请在正文上方注明来源和作者,且不得对内容作实质性改动;微信公众号、头条号等新媒体平台,转载请联系授权。邮箱:shouquan@stimes.cn。