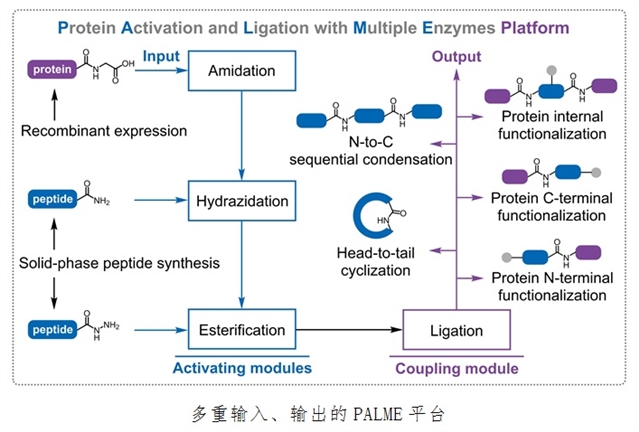
获取与改造蛋白质一直是 领域的核心问题之一,其方法主要有两种:第一是利用生物体内的天然蛋白质合成系统(核糖体),进行蛋白质的重组与表达;第二是近年来蓬勃发展的人工蛋白质合成方法,此类方法以多肽固相合成与特异性拼接为核心,与天然的核糖体系统相比,这些技术所能利用的“分子砌块”不限于天然氨基酸及少数非天然氨基酸,而且能进行各种类型的化学修饰,拓宽了人类在原子水平上人工构筑蛋白质的可能性。然而,目前主流的多肽拼接方法均需设计特殊的连接位点,在连接位点处留下相应的“疤痕”。中科院微生物研究所吴边团队与跨国药企费森尤斯集团合作,在《国家科学评论》(National Science Review, NSR)报道了他们开发的无痕蛋白质酶法合成平台PALME。该平台基于合成生物学理念,对微生物酶资源进行深度挖掘与大尺度计算重塑设计,围绕复杂体系下的碳氮成键反应,串联多个催化元件,以逐级定向活化的方式,使无侧链保护的多肽能够在水相中完成无痕拼接,进而实现完整蛋白质的酶法合成。
PALME平台由上游的活化模块和下游的连接模块组成。该平台对底物序列无特定限制,能够以固相合成多肽、重组表达的天然蛋白质等为输入;仅需在C端引入一个额外甘氨酸,即可由多肽氧化酶、裂解酶和修饰酶共同组成的活化模块对C-端甘氨酸定向无痕活化;活化后的多肽随即被下游的连接模块所识别,在广谱人工多肽连接酶(Peptiligase系列酶)的催化作用下,与另一多肽完成拼接。该平台选用的催化元件具有高度的位置选择性,因此能够合成N/C端修饰蛋白质、中段修饰蛋白质、环状蛋白质等各种结构的蛋白质,为人工蛋白质合成提供全局解决方案。为展示PALME平台的能力,该团队合成了一系列重要的药物多肽与非天然蛋白质。在新型糖尿病药物艾塞那肽的全合成案例中,首次实现了固相合成多肽的N-to-C酶法级联组装。在环化合成实验中,实现了无天然环化酶识别位点的SMAP抗菌肽环化。在功能蛋白的半合成实验中,该团队成功将化学合成片段与重组蛋白质拼装,所获得的半合成异构酶表现出与完整重组酶等效的催化活性。此外,他们还展示了极具挑战的蛋白质双向连接,将化学合成的多肽的两端分别与其他重组蛋白质进行拼装,实现了无自然化学连接位点的乙酰化修饰蛋白质的合成,成功的扩展了蛋白质人工合成的应用空间。近年来,人工智能技术对蛋白质结构预测与功能设计领域产生了颠覆性的影响。合成生物学家开始突破天然氨基酸的限制,将高阶的化学变化引入到蛋白质的设计空间中。该平台为非天然功能蛋白质的构筑提供了工具,为扩展人类设计、合成生物大分子元件的能力提供支撑。
中国科学院微生物研究所高级工程师李瑞峰、博士研究生朱彤,以及费森尤斯的Schmidt博士为该研究的共同第一作者,费森尤斯的Nuijens博士与微生物所吴边研究员为共同通讯作者。该工作的完成还得到微生物研究所方敏研究员与严景华研究员的大力协助。该项研究得到了国家重点研发计划合成生物学专项、国家基金委优青项目、中国科学院先导专项、战略生物资源服务网络计划生物资源衍生库、前沿科学研究计划“从0到1”原始创新项目的支持。(来源:科学网)
相关论文信息:https://doi.org/10.1093/nsr/nwab158