|
|
|
|
|
科学家研发出单细胞和空间转录组中环形RNA深度学习算法 |
|
|
环形RNA是一类广泛表达的非编码RNA,有较高的细胞类型及组织表达特异性,在器官发育及肿瘤发生等过程中起着重要调控作用。环形RNA由3‘端的受体位点和5’端的供体位点共价连接形成,这一过程被称为反向剪接,然而由于缺少poly(A)尾,环形RNA无法被经过polyA富集等转录组建库方式有效捕获。因此,环形RNA的表达信息在绝大部分单细胞以及空间转录组数据中缺失。为了获得丰富的单细胞及空间水平的环形RNA表达信息,需要研发新型环形RNA表达预测算法。
中国科学院北京基因组研究所研究员高远团队与北京
研究院研究员赵方庆团队联合研发了深度学习模型CIRI-deep,以准确预测不同样本间的差异剪接环形RNA。该模型从环形RNA调控机制角度出发,整合了3527个环形RNA特异的顺式元件以及1499个样本特异的反式因子作为输入特征,且不依赖于传统的反向剪接信号识别,可以在任意转录组样本间预测差异剪接的环形RNA。评估结果表明,CIRI-deep可以实现多种转录组测序数据中差异剪接环形RNA的可靠预测,并在单细胞及空间水平实现细胞类型特异环形RNA的准确解析,具有广泛的应用场景。
研究团队从397个深度测序的全转录组样本中识别了超过2500万高度可信的环形RNA差异剪接事件。由于这些剪接事件覆盖了25个人体组织,使用它们作为训练集,使CIRI-deep具有良好的泛化性能。CIRI-deep在测试数据集上的AUROC值达到0.906,并且可以准确预测来自非训练集病理条件与正常样本间的环形RNA差异剪接。此外,在低深度测序的转录本中,CIRI-deep对差异环形RNA的预测效果优于基于reads数的统计检验方法。
为了解释CIRI-deep的预测原理,研究团队研发了深度学习可解释性分析框架Adapted Integrated Gradient,以量化分析组织特异环形RNA的调控因素及其贡献。结果表明,相较于基因序列结构等顺式元件,RNA结合蛋白等反式因子的表达水平对预测准确性的贡献更大,且具有更强的组织特异性。该分析框架验证了已知的环形RNA剪接的调控因素,如剪接位点、内含子区域的Alu元件、FUS蛋白的表达等,也提示了之前未发现的潜在调控因子如NOVA2、KHDRBS3等对环形RNA剪接的影响。
为了从polyA富集测序的单细胞以及空间转录组数据中挖掘环形RNA表达水平差异,研究团队进一步利用polyA数据训练了CIRI-deepA模型。结果表明,CIRI-deepA的预测表现大幅超过直接使用polyA数据推断差异剪接环形RNA的效果。在脑胶质瘤数据集上应用CIRI-deepA,表明该模型可有效预测肿瘤细胞群体和健康细胞群体之间的差异剪接环形RNA。研究团队也将CIRI-deepA应用到了10X单细胞数据集上,准确预测了不同细胞群体的特异高表达环形RNA。另外,在空间转录组数据中,CIRI-deepA可用于预测空间区域特异高表达的环形RNA,并实现对环形RNA表达进行空间区域水平的可视化。利用CIRI-deepA预测的高度特异表达环形RNA,可进一步解析不同区域的细胞类型构成。
CIRI-deep模型可有效用于各转录组样本间推断差异剪接环形RNA,拓展了环形RNA的研究范围,为环形RNA研究提供了新的高效分析方法。同时,CIRI-deepA模型可以提供单细胞及空间水平环形RNA的有效解析,为挖掘细胞类型特异的环形RNA标志物提供了重要的方法学工具。
2月2日,相关成果以CIRI-Deep Enables Single-Cell and Spatial Transcriptomic Analysis of Circular RNAs with Deep Learning为题,发表在《先进科学》(Advanced Science)上。研究工作得到国家重点研发计划、国家自然科学基金等的支持。(来源:中国科学院北京基因组研究所)
相关论文信息:https://doi.org/10.1002/advs.202308115
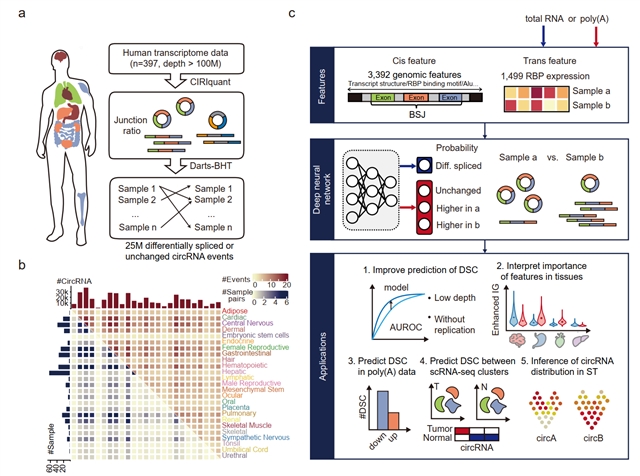
基于深度学习的环形RNA差异剪接预测算法CIRI-deep
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。