|
|
|
|
|
转录因子HNF1A、HNF4A和FOXA2调节肝细胞蛋白N-糖基化 Engineering |
|
|
论文标题: Transcription Factors HNF1A, HNF4A, and FOXA2 Regulate Hepatic Cell Protein N-Glycosylation
期刊:Engineering
发表时间: January 2024
DOI:https://doi.org/10.1016/j.eng.2023.09.019
微信链接:点击此处阅读微信文章
文章速览

来自克罗地亚的科研团队在中国工程院院刊《Engineering》2024年1月刊发表题为“Transcription Factors HNF1A, HNF4A, and FOXA2 Regulate Hepatic Cell Protein N-Glycosylation”(转录因子HNF1A、HNF4A和FOXA2调节肝细胞蛋白N-糖基化)的研究论文。肝细胞核因子1α(HNF1A)、肝细胞核因子4α(HNF4A)和叉头框蛋白A2(FOXA2)是调控肝脏中复杂基因网络的关键转录因子,形成一个转录调控环路。Encode和ChIP-Atlas数据库确定了这些转录因子在许多糖基转移酶基因中的识别位点。该研究结合10个候选糖基因,对HNF1A、HNF4A和FOXA2进行计算机模拟分析,证实这些转录因子在肝脏中显著富集。之前的研究确定了HNF1A是血浆糖蛋白岩藻糖基化、聚糖分支和半乳糖基化的主要调控因子。该研究从功能上验证了这三个转录因子对下游糖基因转录表达的作用以及对聚糖表型的可能影响。使用最先进的规律成簇间隔短回文重复序列/dead CRISPR相关蛋白9(CRISPR/dCas9)分子工具来下调人肝癌细胞系HepG2细胞中的HNF1A、HNF4A和FOXA2基因。研究结果表明,这三个基因的下调单独或成对地影响了许多糖基因的转录活性,尽管糖基因的下调并不总是伴随着相应的聚糖结构的明确变化。这种效应可以被更好地视为总HepG2 N-糖基组的总体变化,主要是由于双触角聚糖的扩展。文章提出了另一种用于评估N-糖基组组成的替代方法,通过量化每个聚糖结构中的单体数量来估计糖基组的整体复杂性。还提出了一个模型,显示了HNF1A-FOXA2和HNF4A-FOXA2的相互激活影响了HepG2细胞中的糖基因和蛋白糖基化。
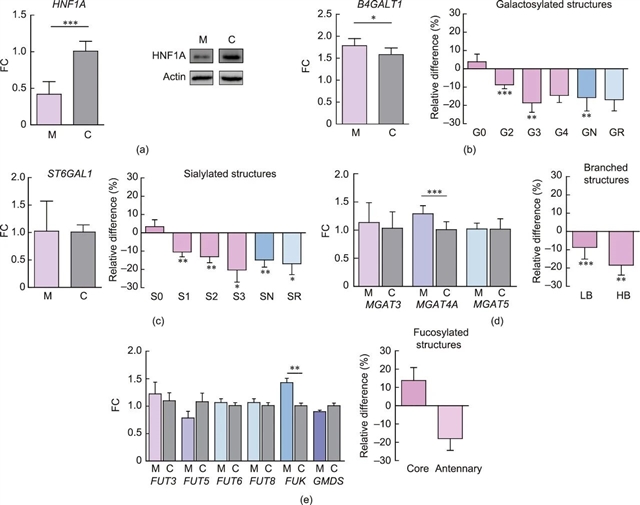
图. 利用KRAB-dSpCas9下调HNF1A对HepG2细胞糖基因转录和总N-糖基组含量的影响。
关键词
规律成簇间隔短回文重复序列,(CRISPR)/死亡CRISPR相关蛋白9(dCas9),表观遗传学,肝细胞核因子1α(HNF1A),肝细胞核因子4α(HNF4A),叉头框蛋白A2(FOXA2),N-糖基化,HepG2细胞
引用信息
Vedrana Vi?i? Bo?kor, Nika Foglar, Goran Josipovi?, Marija Klasi?, Ana Vuji?, Branimir Plavša , Toma Keser , Samira Smajlovi?, Aleksandar Vojta, Vlatka Zoldoš. Transcription Factors HNF1A, HNF4A, and FOXA2 Regulate Hepatic Cell Protein N-Glycosylation. Engineering, https://doi.org/10.1016/j.eng.2023.09.019

Open access
开放获取全文
https://www.engineering.org.cn/engi/EN/10.1016/j.eng.2023.09.019
推荐阅读
北京交通大学团队在智能铁路主动安全保障技术研究获重要进展
中国科学院团队:优化紫外高级氧化工艺,实现水中微污染物精准去除
Engineering“新一代人工智能赋能新型电力系统”国际研讨会在天津大学召开
热管理技术的革新:可重构热穹实现针对带热源目标的三维热隐身
Engineering再次入选中国科技期刊卓越行动计划英文领军期刊项目
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。