|
|
|
|
|
QB 严洪/汤超/赵中应团队合作提出单细胞形态分析应用平台-CShaperApp |
|
|
论文标题: CShaperApp: Segmenting and analyzing cellular morphologies of the developing Caenorhabditis elegans embryo
期刊:Quantitative Biology
作者:Jianfeng Cao, Lihan Hu, Guoye Guan, Zelin Li, Zhongying Zhao, Chao Tang, Hong Yan
发表时间:16 May 2024
DOI:https://doi.org/10.1002/qub2.47
微信链接:点击此处阅读微信文章
自秀丽隐杆线虫(Caenorhabditis elegans)被用作生物研究标本以来,已发展成为发育生物学众多热门研究的重要载体。秀丽隐杆线虫躯体透明、生命周期短且细胞谱系具有高度的重复性,使其培育、观测和分析难度相对降低,成为研究有机生命发育规律的重要窗口。自受精卵到胚胎发生结束,秀丽隐杆线虫包含大约550个活细胞,其中每个细胞均可通过相对位置关系命名和追踪。胚胎发育过程可通过活体荧光显微镜成像捕捉细胞核、细胞膜等不同的细胞结构。然而,显微镜成像质量受到光漂白和光毒性的影响,使得大规模的荧光图像数据信噪比相对较低,这给定量化的细胞形态发育分析带来了巨大挑战。近年来,研究者已提出StarryNite、AceTree等工具,用于识别、追踪并可视化秀丽隐杆线虫胚胎中的细胞核。然而,此类工具难以识别细胞边界,因而在表征细胞形态特征(如细胞形状、细胞体积、细胞表面积以及细胞间接触面积)方面仍然存在不足。尽管已有多项研究提出了用于单细胞水平分割秀丽隐杆线虫胚胎图像分割算法,但目前仍然缺乏交互性的形态分析平台,阻碍了此类算法在生物研究实验室的广泛应用。
近期,香港城市大学严洪课题组、北京大学定量生物学中心汤超课题组和香港浸会大学赵中应课题组联合在Quantitative Biology期刊上发表题为“CShaperApp: Segmenting and analyzing cellular morphologies of the developing Caenorhabditis elegans embryo”的文章,为秀丽隐杆线虫胚胎细胞定量化形态研究提供了高效而精准的分析工具。

全文概要
CShaperApp包含五个模块,即预处理、分割、分析、结果和训练(如图1)。预处理模块将每个时间点的所有显微切片堆叠成一个体积图像(图1A)。当由StarryNite和AceTree生成的细胞核位置和谱系文件可用时,细胞位置信息将用于合成二值细胞核掩模,其中前景像素对应于细胞核中心。分割模块采用CShaper框架对细胞膜荧光图像进行分割,将其转化为数字化的分割结果,清晰识别每个细胞对应的区域(图1B)。分析模块负责自动收集细胞形态特征,如细胞体积、表面积和细胞间接触面积,以Excel格式输出(图1C)。结果模块提供分割后的细胞形态可视化,同时展示所有统计信息。分割后的3D胚胎以不同颜色标记每个细胞(图1D),用户可对统计数据进行质量控制以筛选可靠结果。此外,训练模块允许用户基于特定数据集重新训练模型以获得最佳结果。
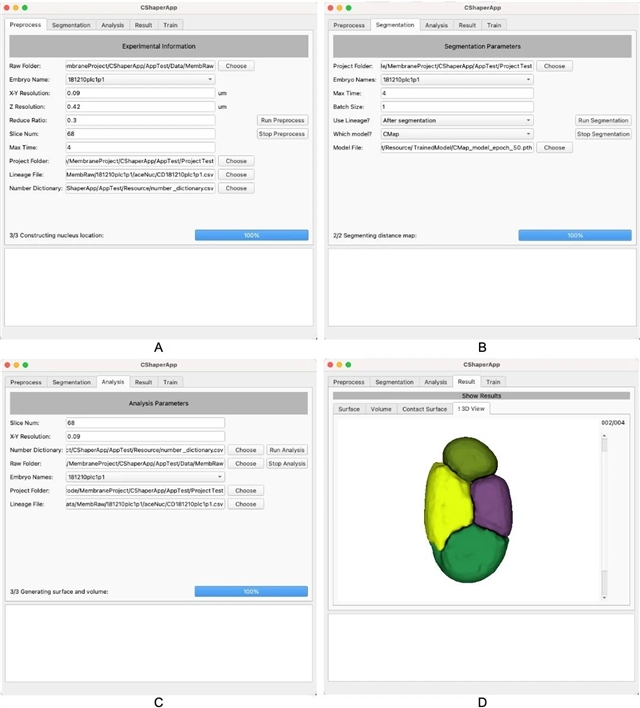
图1. CShaperApp功能展示图,包含预处理(A)、分割(B)、分析(C)和结果展示(D).
为验证分析框架的效率及准确性,CShaperApp在公开数据集spheresDT/Mpacts-PiCS, BCOMS,CShaper上进行了测试。数据集分辨率等差异有利于阐述CShaperApp在不同应用场景中的泛化能力。
1. CShaperApp显著提高形态分析处理效率
来自CShaper的数据集包含150个连续的3D图像,覆盖秀丽隐杆线虫4-细胞至350-细胞阶段,时间分辨率为1.39分钟,其轴向(z)分辨率与横向分辨率(xy)分别为0.09 μm与0.42 μm。分割3D图像时,预处理模块首先将每个时间点的68个切片堆叠;随后将体积图像尺寸从512×712×68调整为153×213×95,获得各向同性空间分辨率0.30 μm。此后应用分割和分析模块获取最终的细胞形态特征。对于配备Intel® Xeon CPU和NVIDIA GPU 3090的工作站,完成所有任务仅需约30分钟,显著缩短了细胞形态研究数据处理时间。
2. 自定义训练模块提升了CShaperApp自适应能力
spheresDT/Mpacts-PiCS数据集的数据可验证CShaperApp是否适用于其他实验室。由于该成像数据的轴向分辨率明显低于CShaper所用的数据(1.20 μm vs 0.42 μm),因而该工作使用CShaperApp中的训练模块,利用spheresDT/Mpacts-PiCS中部分标注微调分割模型,以帮助深度学习模型适应特定的数据分布。如图2所示,重新训练的模型输出了令人满意的结果,展现了CShaperApp在不同实验室中的广泛应用潜力。
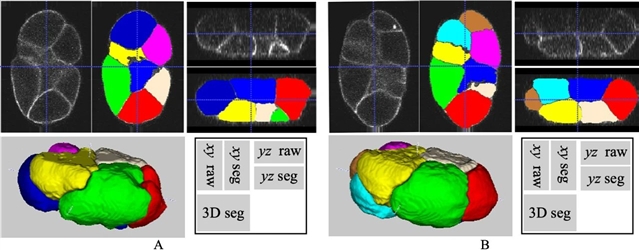
图2. CShaperApp在spheresDT/Mpacts-PiCS数据集上的测试结果.
3. CShaperApp针对不同数据具有鲁棒性
尽管微调深度学习模型是有利的,研究者可能面临缺乏训练用标注数据的问题。此时要求CShaperApp在应用数据发生变化的场景中具备鲁棒性,即保持分割与分析性能。为探究CShaperApp在此方面的能力,我们直接使用CShaperApp预设模型处理BCOMS数据。BCOMS数据轴/轴向分辨率分别为0.50 μm与0.44 μm,其中横向分辨率低于CShaper和spheresDT/Mpacts-PiCS。在未做任何特殊处理的情况下,CShaperApp仍然得到了良好的分割结果(图3)。
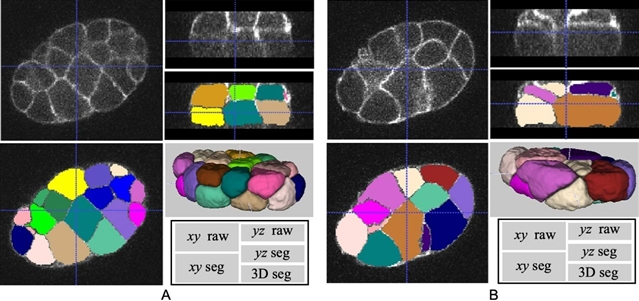
图3. CShaperApp使用预设模型在BCOMS数据集上的测试结果.
4. 未来应用前景
CShaperApp是一款面向发育生物学研究者的高效、可扩展的计算机应用程序,有助于分析胚胎发育中精细的定量细胞形态数据。形态统计结果可为基因特异性表达、细胞靶向凋亡、高通量药物筛选等研究领域提供宝贵的数据支撑,从而服务于癌症治疗等临床应用。同时,得益于模块化框架的灵活性,CShaperApp可通过将分割模型替换为更先进的算法而实现优化,进一步促该领域的发展。
QB期刊介绍
Quantitative Biology (QB)期刊是由清华大学、北京大学、高教出版社联合创办的全英文学术期刊。QB主要刊登生物信息学、计算生物学、系统生物学、理论生物学和合成生物学的最新研究成果和前沿进展,并为
与计算机、数学、物理等交叉研究领域打造一个学术水平高、可读性强、具有全球影响力的交叉学科期刊品牌。
《前沿》系列英文学术期刊
由教育部主管、高等教育出版社主办的《前沿》(Frontiers)系列英文学术期刊,于2006年正式创刊,以网络版和印刷版向全球发行。系列期刊包括基础科学、
、工程技术和人文社会科学四个主题,是我国覆盖学科最广泛的英文学术期刊群,其中12种被SCI收录,其他也被A&HCI、Ei、MEDLINE或相应学科国际权威检索系统收录,具有一定的国际学术影响力。系列期刊采用在线优先出版方式,保证文章以最快速度发表。
中国学术前沿期刊网
http://journal.hep.com.cn

特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。