论文标题:3D genomic organization in cancers(癌症三维基因组研究进展)
期刊:Quantitative Biology
作者:Junting Wang, Huan Tao, Hao Li, Xiaochen Bo, Hebing Chen
发表时间:03 Nov 2022
DOI:10.15302/J-QB-022-0317
微信链接:点击此处阅读微信文章
癌症是一种死亡率很高的恶性疾病,严重危害着人类健康。广义上讲,癌症是肿瘤细胞持续恶性增殖的产物,具有自给自足的生长信号、对生长抑制信号不敏感、逃避细胞凋亡、无限复制的潜力、持续的血管新生及组织侵袭转移等特征,这些特征使细胞从正常状态转变为肿瘤生长状态。
染色质三维结构在细胞分化、衰老和转录调控等生物过程中发挥着重要作用,其结构改变存在于多种癌症中,然而其潜在机制尚不完全清楚。近日,军事医学研究院伯晓晨教授和陈河兵教授团队在Quantitative Biology上发表了题为“3D genomic organization in cancers”的综述文章。文章详细介绍了染色质三维结构、三维基因组在癌症发生、发展中的作用以及用于探索癌症三维基因组的人工智能算法。

全文概要
癌症的发生、发展往往伴随着三维基因组的结构改变,染色质三维结构畸变扰乱基因表达是正常细胞发生致癌转变的主要驱动力之一。染色质拓扑结构的变化会改变基因的调控环境,最终影响基因表达,推动癌症的发生、发展。基因突变使染色质三维结构发生畸变,导致细胞发生致癌转变。鉴于染色质三维结构在基因表达中的重要调控作用,研究其在癌症中的作用将有助于了解癌症发生、发展的分子机制。
本文总结了癌症中不同层级染色质三维结构的变化,以及这些变化是如何驱动了癌症的发生、发展。从染色质高级结构的角度理解癌症发生的内在机制,将为癌症的精确诊断和个性化治疗提供新的见解。此外,本文综述了用于识别染色质三维结构和增强Hi-C数据分辨率的人工智能(artificial intelligence,AI)模型,这些模型的应用进一步揭示了三维基因组在癌症发展中的独特作用。
癌症三维基因组图谱
近年来,伴随着以高通量染色体构象捕获(high-throughput chromosome conformation capture,Hi-C)为代表的三维基因组解析技术的蓬勃发展,三维基因组在癌症发生、发展中扮演的角色被逐步发掘(如图1)。
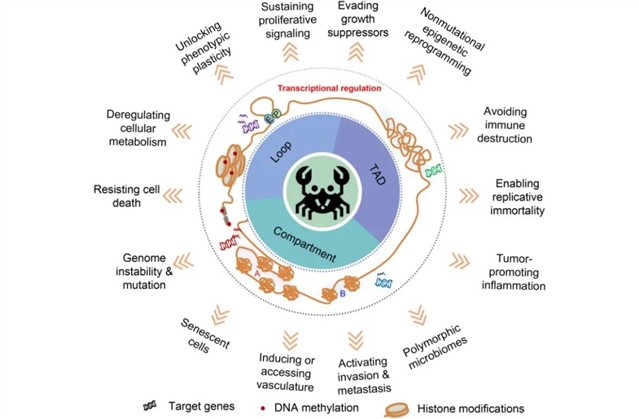
图1. 癌症三维基因组图谱。细胞的染色质三维结构改变影响靶基因的表达使细胞获得癌症表型。
染色质的多层级空间结构
随着高通量染色体构象捕获,配对标签测序(chromatin interaction analysis by paired-end tag sequencing,CHIA-PET)和以蛋白为中心的染色质构象分析(proteincentric chromatin conformation assay,HiChIP)等技术的出现,染色质的多层级空间结构被逐步解析:细胞核中的每条染色体占据一个相对独立的区域,即染色体疆域(chromosome territory)(如图2A,图2B)。染色体疆域又可分为A区室(compartment)和B区室,分别对应着染色质的活性和非活性状态(如图2C),从A区室到B区室的转换(switch)与基因表达的下调相关。A/B区室被分离成百万碱基大小的拓扑关联结构域(topologically associated domain,TAD),TAD内部由染色质环(chromatin loop)构成(如图2D和2E)。TAD边界由CTCF(CCCTC-binding factor)、黏连蛋白(cohesin)分隔,同一TAD内的基因在细胞分化过程中往往是共表达的。当TAD游离到细胞核的边缘时,其内部基因的表达被抑制,当其位于细胞核中心区域时,TAD内基因表达变得活跃。染色质环是染色质空间结构调控的基本单位,大多形成于TAD内部的增强子和启动子间,是基因表达所必需的。

图2.多层级染色质三维结构。(A)细胞核,(B)染色质疆域,(C)染色质区室,(D)拓扑关联结构域,(E)染色质环。黄色的点代表转录因子CTCF,红色、蓝色箭头代表CTCF的结合方向,橘黄色的环代表黏连蛋白复合物。
癌症中的区室转换
2009年,Aiden等用Hi-C技术研究了人类淋巴细胞的三维结构,并提出了A/B区室的概念(如图3)。随后,Barutcu等发现了A/B区间的转换,揭示了正常细胞和乳腺癌细胞间A/B区室转换与相应基因的表达变化有关。2017年,北京大学李程课题组在骨髓瘤样本中观察到了区室转换,他们发现区室转换会影响骨髓瘤相关基因的表达。近期,研究者又在结直肠腺瘤中观察到了I区室(intermediate compartment),I区室可以转化为细胞类型特异的A区室或B区室。正常细胞中,I区室接近于A区室,通常表现为低甲基化;而在癌细胞中,I区室更接近于B区室,表明广泛的区室重塑可能与肿瘤抑制作用相关。Blasi等在不同的慢性淋巴细胞白血病和套细胞淋巴瘤亚型中发现了高度动态的I区室,其启动子区域富集了H3K27me3。疾病特异的染色质区室结构变化往往涉及广泛的基因转录激活,与癌症发生密切相关。
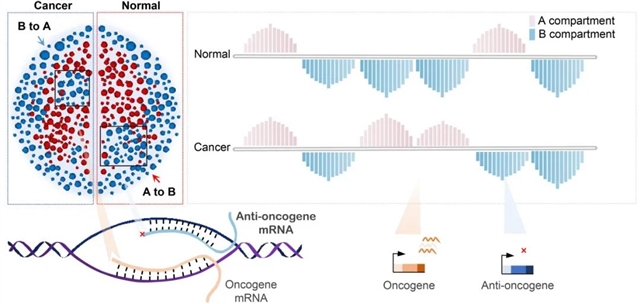
图3.染色质A/B区室转换通常伴随着基因表达活性增强或抑制。粉色:A区室,蓝色:B区室,条形图的高度代表信号强度。不同颜色代表不同基因的转录活性。
癌症中的TAD改变
TAD将基因组分成~Mb的富含CTCF蛋白边界的区域(如图4)。它的形成主要是由染色质分隔和环状挤压驱动的。TAD通常由绝缘子隔开,一个TAD内包含一个或几个基因以及它们的启动子、增强子,形成独立的调控单元。三维染色质结构变异会影响TAD的完整性和增强子-启动子的相互作用,从而影响基因表达,导致人类疾病。癌症中基因组突变位点累积十分常见,而癌症基因组中体细胞突变的分布与染色质三维结构相关,体细胞突变频率在TAD边界更为明显。
随着TAD预测算法的发展,研究人员发现了比传统TAD尺寸更小的亚TAD。基因表达调控核心复合物LDB1基因位于PU.1附近75kb,保持了粒细胞分化过程中亚TAD结构的稳定性,而LDB1敲除会破坏PU.1所在亚TAD的稳定性。特定情况下,衰老细胞通过衰老相关分泌表型促进癌症的发生、发展,而亚TAD重组被认为是驱动细胞脱离癌基因引发的衰老,获得侵袭性特征的起始因素。
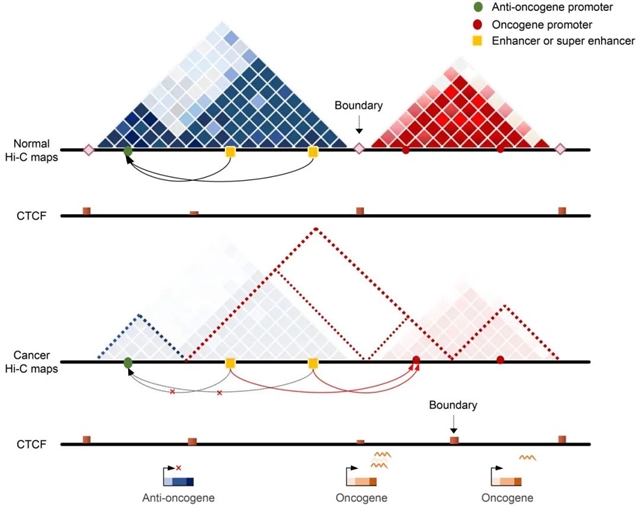
图4.癌症中TAD或亚TAD的重组导致的增强子劫持。Hi-C图谱显示了正常细胞与癌症细胞中的TAD或亚TAD重组。绿色、红色的圆点分别代表抑癌基因和癌基因的启动子,黄色的方块代表增强子或超级增强子,菱形代表TAD边界,红色条形图代表的是CTCF的信号值,黑色箭头代表正常细胞中的相互作用,红色箭头代表癌细胞中新产生的相互作用。
癌症中的染色质环异常
染色质环调控远端调控元件,如增强子和启动子的相互作用,以调控基因表达。染色质环的异常与癌症密切相关,如在不同亚型的急性髓系淋巴细胞白血病研究中发现,增强子劫持是染色质环异常致癌的重要机制(如图5)。据报道,前列腺癌细胞中雄性激素受体位点的染色质结构改变,形成了癌症特异性增强子-启动子环。急性淋巴细胞白血病细胞的相分离结构表明,嵌合型肿瘤蛋白NUP98-HOXA9诱导癌基因形成异常染色质环,导致癌基因激活增强。黑色素瘤中STAG2的调控模式揭示了H3K27ac相关的染色质环增强使IRF9和PD-L1表达上调,促进STAG2突变肿瘤细胞的免疫逃避。这从癌症三维基因组角度阐明了癌细胞是如何通过适应性免疫抵抗来实现免疫逃避和自我保护的。

图5.异常的增强子-启动子环激活癌基因的表达。染色体结构变异(例如,染色体易位)会调控增强子-启动子环的形成,影响基因表达并驱动癌症的发生、发展。深蓝色和红色条形代表不同的染色体,黄色条形代表染色体易位区域,绿色和紫色条形分别代表抑癌基因和癌基因启动子,红色圆点代表增强子,黄色圆点代表CTCF,红色和蓝色箭头分别代表CTCF的结合方向。
癌症三维基因组解析工具
近年来,基于机器学习和深度学习的AI模型在染色质三维结构解析领域具有广泛应用(如图6)。这些AI模型可以识别多层级染色质三维结构,如染色质区室、TAD和染色质环等,还能增强Hi-C数据的分辨率。人工智能的应用极大地促进了癌症三维基因组的研究。探索癌症中染色质三维结构的改变,能够进一步完善癌症三维基因组的调控图谱。

图6.基于AI的多组学数据整合促进了癌症研究。基于AI整合癌症多组学数据有助于深入理解癌症基因组的发生、发展机制,寻找癌症治疗方法。
染色质区室的识别工具
早期染色质区室的识别方法主要是基于主成分分析(Principal Component Analysis,PCA)的降维方法,如Juice-Box、HOMER和Fan-C,然而使用PCA降维的计算速度较慢。为了解决这些问题,Zheng等开发了基于Hi-C数据识别A/B区室的统计学模型——CscoreTool,其计算速度和内存效率较之前提升了30倍。随后识别能力更强的染色质区室识别工具逐步发展,例如马坚教授课题组研发的SNIPER和刘元龙博士等研发的Calder可以基于中等分辨率的Hi-C数据识别亚区室,Pentad能够识别染色质区室中接触频率的重新分布,并评估区室强度的改变。
TAD的识别工具
2018年,清华大学陈阳和张奇伟课题组合作提出了HiCDB,它能够基于局部相对绝缘系数和多尺度聚集方法来计算TAD的边界。OnTAD基于评分递归的动态算法,可以用一系列大小不同的滑动窗口扫描基因组来识别TAD边界,并将识别出的边界经优化组装成多层级TAD结构。TARGET可以识别肿瘤中特异性改变的TAD边界,并预测肿瘤中受TAD异常改变调控的异常表达基因。开放染色质空间密度(The spatial density of the open chromatin,SDOC)是近年来提出的TAD内定量指标,用来反映TAD内的表观遗传和基因转录特征。SDOC能够识别边界没有变化,而内部密度改变的TAD结构,为发现致癌性转化的转录调控机制奠定了基础。
Loop的识别工具
为了探究染色质环在癌症发生、发展中的调控模式,岳峰课题组提出了基于Hi-C数据鉴定癌症基因组的增强子劫持的工具NeoLoopFinder。该工具通过矫正拷贝数变异、SV杂合性和异质性带来的数据偏倚,来识别SV介导的染色质环,该工具目前被广泛应用于膀胱癌和脑胶质瘤的研究中。Cameron等提出了基于相邻片段之间的依赖关系的Hi-C交互频率推断(Hi-C Interaction Frequency Inference,HIFI)算法。FitHiC1/2能够基于统计置信度估计来检测染色质环。3DPredictor基于CTCF结合信号和基因表达来定量预测染色质互作。中南大学李敏课题组研发的LoopPredictor可用于预测由增强子介导的全基因组互作,并能够从肿瘤细胞系中分离出细胞类型特异性的基因调控网络。近期开发的EPIXplorer能够预测远距离增强子-启动子互作,有助于解析全基因组关联研究中发现的变异是如何影响癌进展的。
Hi-C数据的增强工具
目前深度学习和机器学习已被越来越多地应用于Hi-C数据增强,有助于癌症三维基因组的探索。HiCPlus使用深度卷积神经网络提高Hi-C数据分辨率,是在Hi-C数据增强方面应用深度学习的先驱。随后,基于深度学习的Hi-C数据增强方法不断涌现,如HiCNN1/2、hicGAN和DeepHiC,其中hicGAN和DeepHiC都是基于生成式对抗网络的工具。此外,DeepHiC提供了用户友好的网络交互界面,只需要几个简单的步骤就可以实现Hi-C数据的增强。
结论和展望
随着癌症染色质三维结构的深入研究,我们对癌症的发生、发展机制有了更全面的了解。染色质三维结构,如A/B区室、TAD和染色质环,它们动态联系、协同调控癌基因的表达,从而调控癌症进程。近年来,基于AI的计算工具逐步应用于染色质三维结构的识别和Hi-C数据增强,使癌细胞中染色质三维结构的识别更加准确、精细,有利于探索癌症三维基因组的进一步探索。
尽管癌症三维基因组学发展迅速,但目前的研究仍存在很多局限性。首先,传统的Hi-C数据仅反映了细胞群的平均特征,无法完全揭示肿瘤的异质性。相比之下,单细胞Hi-C数据可以解析单个细胞的染色体构象,并阐明染色体相互作用和基因组功能的调节机制,但高昂的成本和繁琐的数据处理过程限制了其广泛应用。癌症多组学数据的不断产生和AI的发展,促进了癌症研究从低维到高维的转变(如图7A和7B),以及多组学数据的处理和整合,促进了肿瘤发生、发展、诊断、治疗和预后的研究(图7B和7C)。此外,现有方法仍然缺乏可解释性。可解释AI模型的发展使计算模型更加透明、逻辑性更强,能够促进海量测序数据的进一步整合,可解释AI在肿瘤多组学数据处理中的应用将进一步揭开癌症精准治疗的面纱。
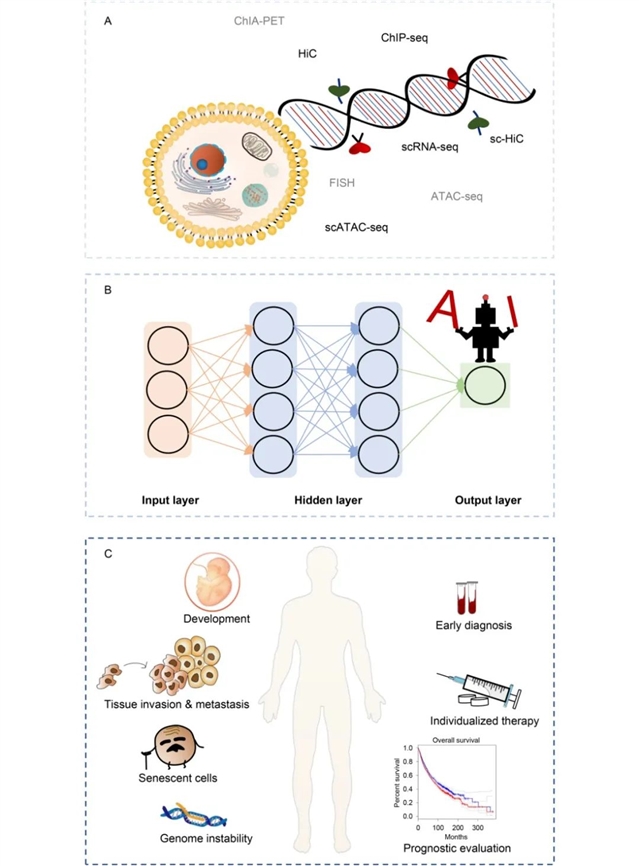
图7.癌症发生机制和治疗方法研究的新机遇。(A)多组学数据整合,(B)AI算法的发展,(C)AI在癌症发生机制和治疗研究中的应用前景。
从浩瀚的宇宙到微小的细胞,人类在不断认识和改造世界。我们对人类基因组的探索仍处于初始阶段,但我们坚信癌症基因组的调控机制会得到更全面的阐明。相信未来的某一天,癌症不再是不治之症。
致谢
军事医学研究院硕士研究生王军婷和陶欢为该论文的第一作者,军事医学研究院伯晓晨研究员和陈河兵副研究员为该论文的共同通讯作者,北京大学李程研究员对该文章提出了宝贵的指导意见。
QB期刊介绍
Quantitative Biology (QB)期刊是由清华大学、北京大学、高教出版社联合创办的全英文学术期刊。QB主要刊登生物信息学、计算生物学、系统生物学、理论生物学和合成生物学的最新研究成果和前沿进展,并为 与计算机、数学、物理等交叉研究领域打造一个学术水平高、可读性强、具有全球影响力的交叉学科期刊品牌。
QB期刊目前已被ESCI, Scopus, CSCD等国内外重要数据库收录。Citescore2021=4.6,2023年将获得第一个影响因子(IF)。

《前沿》系列英文学术期刊
由教育部主管、高等教育出版社主办的《前沿》(Frontiers)系列英文学术期刊,于2006年正式创刊,以网络版和印刷版向全球发行。系列期刊包括基础科学、 、工程技术和人文社会科学四个主题,是我国覆盖学科最广泛的英文学术期刊群,其中13种被SCI收录,其他也被A&HCI、Ei、MEDLINE或相应学科国际权威检索系统收录,具有一定的国际学术影响力。系列期刊采用在线优先出版方式,保证文章以最快速度发表。
中国学术前沿期刊网
http://journal.hep.com.cn

特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。