近年来
伪狂犬、猴痘等病毒
从动物向人类外溢
引发新发传染病甚至全球大流行
的频率正在显著增加
如何精准预测和预报动物源新发传染病是关系绿色健康养殖与公共卫生防控的重要科学问题
复旦大学公共卫生学院
粟硕教授团队与合作者
运用前沿交叉研究方法
揭示多种哺乳动物宏基因组数据中的
病毒基因组组成、生态学特征
与跨物种传播规律
在“同一健康”理念下
为构建人-动物-环境一体化
多维度新发传染病预测预警体系
奠定重要数据基础
成果于北京时间9月4日以题为
“Farmed fur animals harbor viruses
with zoonotic spillover potential”的研究长文
在《自然》杂志(Nature)发表

聚焦养殖哺乳动物
发现潜在“风险”病毒
21世纪以来,人类多次经历由动物源新发传染病原引发的大流行疫情。2014年西非埃博拉病毒疫情、2015年寨卡病毒在南美暴发并蔓延全球,以及近期起源非洲的猴痘疫情等,接连不断地给全球公共卫生体系带来严峻挑战。
据报道,超过70%的人畜共患疾病源自野生动物。随着全球城市化进程加速,密集的城郊人口、集约化养殖、便捷旅行及环境变化等社会生态因素促使新发传染病频发。这些传染病常由动物引发,并在人畜密切接触、非洲南美洲一些卫生条件不佳的地区显现,公共卫生防线前置需求迫在眉睫。
尽管先前大量碎片化的研究提示部分养殖哺乳动物具有携带人兽共患病毒的潜力(https://pubmed.ncbi.nlm.nih.gov/28284614/),但哺乳动物种类多样,除传统家畜外,缺乏对其他养殖哺乳动物多地域、多类群、跨物种等不同维度内的综合病毒组挖掘与跨尺度信息整合研究。当前这些动物的病原生态学与流行病学数据存在较大空白,制约了新发传染病疫情的精准防控。
“我们的研究希望填补当前对动物携带微生物的认知空白,尤其是养殖哺乳动物。”粟硕表示,大多数人类病毒都源自动物传播,但究竟哪些动物携带何种病毒、这些病毒未来是否会对人类构成潜在威胁,目前仍存在较大不确定性。“如果能提前监测和预报这些病毒,并部署包括疫苗研发在内的综合防控措施,这对于新发传染病关口前移、保障人类健康至关重要。”
团队对来自5个动物目的哺乳动物进行了系统的宏基因组研究,鉴定了125种病毒基因组,其中39种可推定为新的病毒种,丰富了养殖哺乳动物携带病毒的种类(图1)。使用生态学地理分布、进化动力学、公共大数据等多学科交叉方法,团队遴选出诺如病毒、盖塔病毒等多种频繁发生“宿主跳跃”的潜在“风险”病毒,解析其跨物种传播规律,并从空间聚类、动物类群、种群、组织等多维度揭示了潜在“风险”病毒的生态学与流行病学特征。
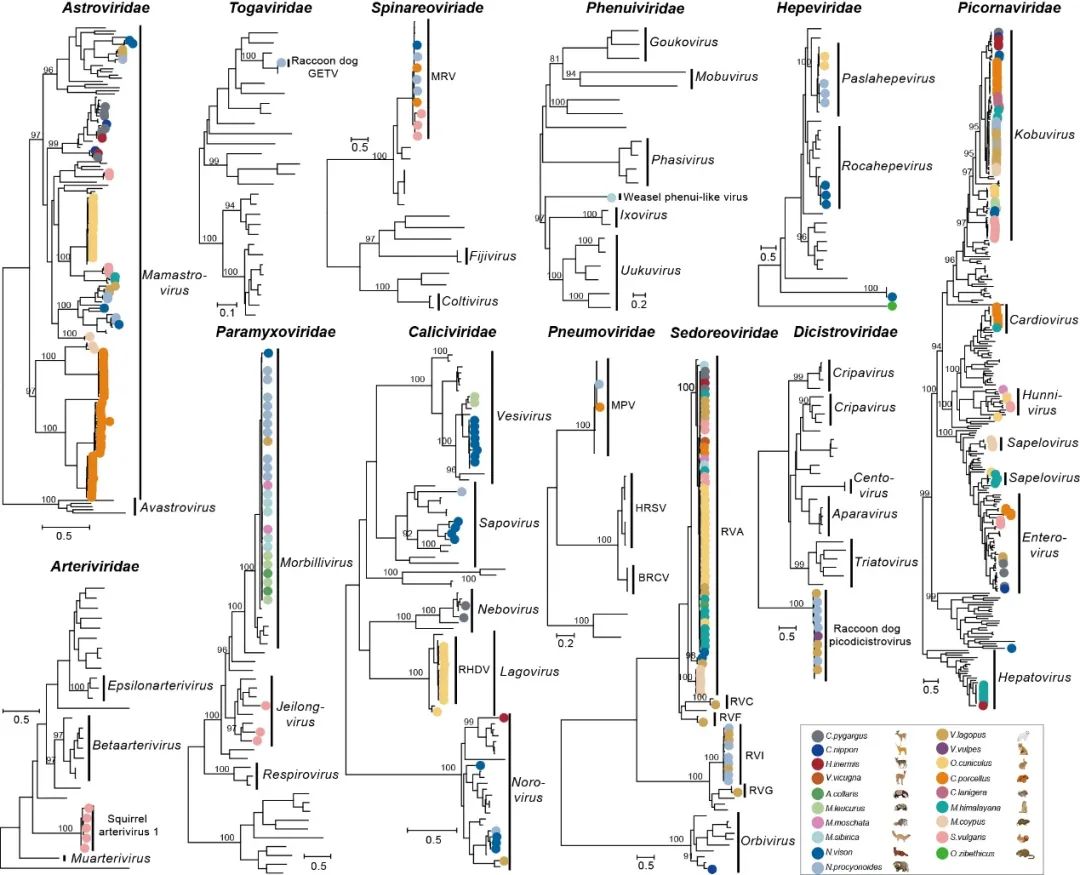
图1:哺乳动物携带RNA病毒的多样性
多学科交叉
寻找病毒检测分析新方法
下一代测序技术的成熟推动了全球基因组学的挖掘与共享进程,在既往的组学研究中,研究人员通常按物种或采样地点进行混样,从而初步解析病毒多样性。然而,传统的混合样本测序无法清晰揭示个体之间或生态系统中不同组成部分之间的关系,还存在样本主要集中于粪便、难以区分环境污染等问题。此外,低丰度病毒面临基因组碎片化、片段化的挑战,混合样本难以实现病毒片段的精准分类,大大制约了病毒基因组的获取。
针对这些局限,团队设计了单组织单样本的建库策略,实现了在个体水平解析病毒共感染以及更灵敏的挖掘低丰度病毒。研究在166个单组织文库里鉴定到2种以上的病毒,发现星状病毒、细小病毒等与其他病毒频繁发生共感染。通过对低丰度病毒片段的精准拼接,避免了真阳性病毒因丰度阈值而被过滤,实现了多个病毒的90%基因组完成度(图2)。
“我们采用的新方法可以精确地在个体层面解析病毒共感染状况,并能更灵敏地检测低丰度病毒,揭示出动物组织器官、个体乃至群体间的多层次病原分布情况,从而获得更为丰富和高分辨率的数据。”粟硕介绍。这意味着研究人员能获得更加细致的分析结果、进行更深入的统计学分析,从而揭示不同尺度上的病原生态学问题。
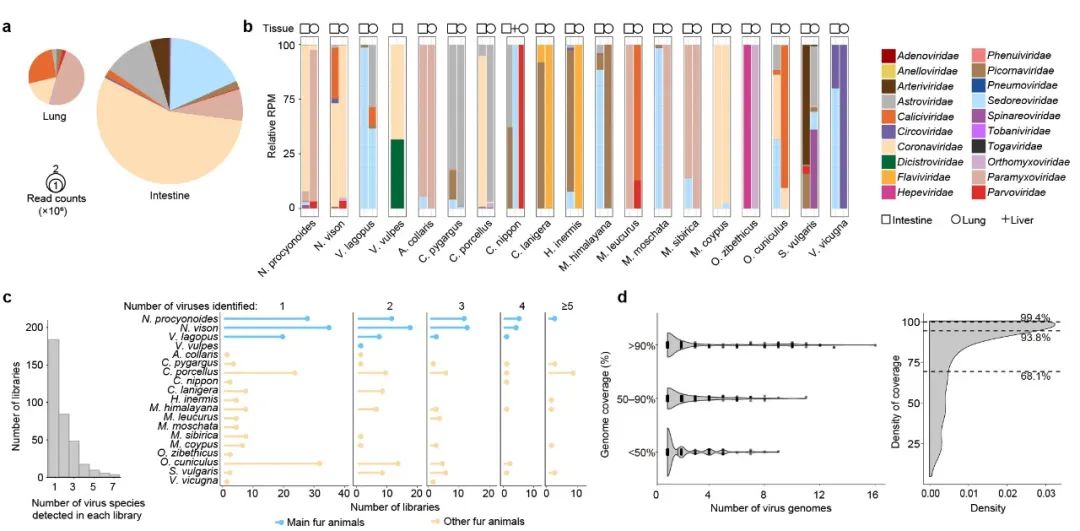
图2:哺乳动物的高分辨率病毒组
研究团队的跨学科交叉研究方法,为其他新发病原体的风险评估树立了范例,为构建多维度新发传染病风险评估体系奠定了基础。这不仅对公共卫生领域产生深远影响,同时也为病毒学、生态学等相关领域提供了新的研究视角和方法指引。
在将近三年的研究过程中,团队面临的最大挑战在于海量宏基因组数据的处理与分析。“分析这些数据需要过硬的生物信息学能力。我们要从海量的原始数据中筛选出病毒信号,并确定哪些是真正的病毒序列。”粟硕带领团队构建高效的一体化宏病毒组注释管道,用于数据清洗、病毒识别和生物信息学分析,成功攻克这一难题。
丰富病毒本底数据,为新发病毒预测预警提供支撑
研究获得的病毒数据,丰富了哺乳动物的病毒本底数据,通过揭示养殖哺乳动物体内病毒的复杂组成,探索病毒跨物种传播的潜力,该研究强调了将公共卫生安全防线前移的重要性。
“现阶段潜在风险病原体并没有公共卫生风险,但对于那些在动物中已经表现出较高流行趋势的病原体,应当加强其在动物中的监测和防控。”粟硕表示,盖塔病毒已在我国多种养殖和野生动物中被监测到,具有频繁跨物种传播能力,应加强对盖塔病毒的监测并尽早储备动物疫苗,保障动物健康养殖以及预防其潜在跨物种传播风险。
近年来,复旦大学粟硕团队致力于应用多学科交叉技术进行新发病毒综合防控研究。此前团队已经在《细胞》(Cell)杂志发表多篇论文,揭示了哪些动物携带和可能传播风险病原,讨论了针对公共卫生防控在未来可采用的宏基因组和多种交叉研究全新监测方法。2024年8月,团队应邀在Cell上发表对全球高致病性禽流感A(H5N1)疫情防控的观点(https://www.cell.com/cell/abstract/S0092-8674(24)00784-0)。文章针对H5N1在美国奶牛场暴发且出现突变病毒(PB2-E627K)外溢到四名奶牛场工人的案例,强调了养殖动物是除禽类以外的人类感染的潜在前线,提出了在环境-动物-人类界面上跨学科研究、全景监测防控禽流感病毒的前沿观点。

图3:课题组研究生努力突破研究瓶颈
研究团队年轻而富有活力,论文的前三位共同第一作者均是课题组在读研究生。下一步,团队将继续聚焦于人和动物新发病毒的预测预报和综合防控研究,将病毒宏基因组等新技术与传统的流行病学数据、环境和生态因素相结合,并针对研究瓶颈按需求开发新的生物信息分析方法,力争为传染病防控和公共卫生安全提供更有力的科学支撑。
原文链接:
https://www.nature.com/articles/s41586-024-07901-3
(原题:宏基因组研究新突破!复旦Nature+1)
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。